JavaScript is disabled for your browser. Some features of this site may not work without it.
Navegação por autor "10753"
-
SUZUKI, MIRIAM F.
 ; OLIVEIRA, JOAO E.
; OLIVEIRA, JOAO E.
 ; DAMIANI, RENATA; LIMA, ELIANA R.
; DAMIANI, RENATA; LIMA, ELIANA R.
 ; AMARAL, KLEICY C.
; AMARAL, KLEICY C.
 ; SANTOS, ANDERSON M. de S.; MAGALHÃES, GERALDO S.; FAVERANI, LEONARDO P.; PEREIRA, LUIS A.V.D.; SILVA, FABIANA M.; BARTOLINI, PAOLO
; SANTOS, ANDERSON M. de S.; MAGALHÃES, GERALDO S.; FAVERANI, LEONARDO P.; PEREIRA, LUIS A.V.D.; SILVA, FABIANA M.; BARTOLINI, PAOLO

.
Human bone morphogenetic protein‑2 (hBMP‑2) characterization by physical–chemical, immunological and biological assays.
AMB Express,
v. 10,
n. 1,
p. 1-10,
2020.
DOI:
10.1186/s13568-020-0964-5
Abstract:
Commercially available preparations of methionyl-human BMP-2 and CHO-derived hBMP-2, which belongs to the
transforming growth factor β (TGF-β) superfamily, were used for a complete characterization. This protein is an
extremely efficient osteoinductor that plays an important role during bone regeneration and embryonic development.
Characterization was carried out via SDS-PAGE and Western blotting, followed by reversed-phase HPLC, sizeexclusion
HPLC and MALDI-TOF-MS. The classical in vitro bioassay, based on the induction of alkaline phosphatase
activity in C2C12 cells, confirmed that hBMP-2 biological activity is mostly related to the dimeric form, being ~ 4-fold
higher for the CHO-derived glycosylated form when compared with the E. coli counterpart. The E. coli-derived methBMP-
2 has shown, by MALDI-TOF-MS, a large presence of the bioactive dimer. A more complex molecular mass
(MM) distribution was found for the CHO-derived product, whose exact MM has never been reported because of its
variable glycosylation. A method based on RP-HPLC was set up, allowing a quantitative and qualitative hBMP-2 determination
even directly on ongoing culture media. Considering that hBMP-2 is highly unstable, presenting moreover
an extremely high aggregate value, we believe that these data pave the way to a necessary characterization of this
important factor when synthesized by DNA recombinant techniques in different types of hosts.
Palavras-Chave:
proteins;
skeleton;
human populations;
bone cells;
cho cells;
bioassay;
escherichia coli;
connective tissue cells
SUZUKI, MIRIAM F.; OLIVEIRA, JOAO E.; DAMIANI, RENATA; LIMA, ELIANA R.; AMARAL, KLEICY C.; SANTOS, ANDERSON M. de S.; MAGALHÃES, GERALDO S.; FAVERANI, LEONARDO P.; PEREIRA, LUIS A.V.D.; SILVA, FABIANA M.; BARTOLINI, PAOLO.
Human bone morphogenetic protein‑2 (hBMP‑2) characterization by physical–chemical, immunological and biological assays.
AMB Express,
v. 10,
n. 1,
p. 1-10,
2020.
DOI:
10.1186/s13568-020-0964-5.
Disponível em: http://repositorio.ipen.br/handle/123456789/31104. Acesso em: $DATA.
Esta referência é gerada automaticamente de acordo com as normas do estilo IPEN/SP (ABNT NBR 6023) e recomenda-se uma verificação final e ajustes caso necessário.
Como referenciar este item
-
LIMA, ELIANA R.
 ; FREIRE, RENAN P.
; FREIRE, RENAN P.
 ; SUZUKI, MIRIAM F.
; SUZUKI, MIRIAM F.
 ; OLIVEIRA, JOAO E.
; OLIVEIRA, JOAO E.
 ; YOSIDAKI, VANESSA L.
; YOSIDAKI, VANESSA L.
 ; PERONI, CIBELE N.
; PERONI, CIBELE N.
 ; SEVILHANO, THAIS
; SEVILHANO, THAIS
 ; ZORZETO, MOISES; TORATI, LUCAS S.; SOARES, CARLOS R.J.
; ZORZETO, MOISES; TORATI, LUCAS S.; SOARES, CARLOS R.J.
 ; LIMA, IGOR D. de M.; KRONENBERGER, THALES; MALTAROLLO, VINICIUS G.; BARTOLINI, PAOLO
; LIMA, IGOR D. de M.; KRONENBERGER, THALES; MALTAROLLO, VINICIUS G.; BARTOLINI, PAOLO

.
Isolation and characterization of the Arapaima gigas growth hormone (ag-GH) cDNA and three-dimensional modeling of this hormone in comparison with the human hormone (hGH).
Biomolecules,
v. 13,
n. 1,
p. 1-17,
2023.
DOI:
10.3390/biom13010158
Abstract:
In a previous work, the common gonadotrophic hormone α-subunit (ag-GTHα), the ag-FSH β- and ag-LH β-subunit cDNAs, were isolated and characterized by our research group from A. gigas pituitaries, while a preliminary synthesis of ag-FSH was also carried out in human embryonic kidney 293 (HEK293) cells. In the present work, the cDNA sequence encoding the ag-growth hormone (ag-GH) has also been isolated from the same giant Arapaimidae Amazonian fish. The ag-GH consists of 208 amino acids with a putative 23 amino acid signal peptide and a 185 amino acid mature peptide. The highest identity, based on the amino acid sequences, was found with the Elopiformes (82.0%), followed by Anguilliformes (79.7%) and Acipenseriformes (74.5%). The identity with the corresponding human GH (hGH) amino acid sequence is remarkable (44.8%), and the two disulfide bonds present in both sequences were perfectly conserved. Three-dimensional (3D) models of ag-GH, in comparison with hGH, were generated using the threading modeling method followed by molecular dynamics. Our simulations suggest that the two proteins have similar structural properties without major conformational changes under the simulated conditions, even though they are separated from each other by a >100 Myr evolutionary period (1 Myr = 1 million years). The sequence found will be used for the biotechnological synthesis of ag-GH while the ag-GH cDNA obtained will be utilized for preliminary Gene Therapy studies.
Palavras-Chave:
fishes;
pituitary hormones;
gonadotropins;
molecules;
cloning;
dna sequencing
LIMA, ELIANA R.; FREIRE, RENAN P.; SUZUKI, MIRIAM F.; OLIVEIRA, JOAO E.; YOSIDAKI, VANESSA L.; PERONI, CIBELE N.; SEVILHANO, THAIS; ZORZETO, MOISES; TORATI, LUCAS S.; SOARES, CARLOS R.J.; LIMA, IGOR D. de M.; KRONENBERGER, THALES; MALTAROLLO, VINICIUS G.; BARTOLINI, PAOLO.
Isolation and characterization of the Arapaima gigas growth hormone (ag-GH) cDNA and three-dimensional modeling of this hormone in comparison with the human hormone (hGH).
Biomolecules,
v. 13,
n. 1,
p. 1-17,
2023.
DOI:
10.3390/biom13010158.
Disponível em: http://repositorio.ipen.br/handle/123456789/33977. Acesso em: $DATA.
Esta referência é gerada automaticamente de acordo com as normas do estilo IPEN/SP (ABNT NBR 6023) e recomenda-se uma verificação final e ajustes caso necessário.
Como referenciar este item
-
FREIRE, RENAN P.
 ; HERNANDEZ-GONZALEZ, JORGE E.; LIMA, ELIANA R.
; HERNANDEZ-GONZALEZ, JORGE E.; LIMA, ELIANA R.
 ; SUZUKI, MIRIAM F.
; SUZUKI, MIRIAM F.
 ; OLIVEIRA, JOAO E. de
; OLIVEIRA, JOAO E. de
 ; TORATI, LUCAS S.; BARTOLINI, PAOLO
; TORATI, LUCAS S.; BARTOLINI, PAOLO
 ; SOARES, CARLOS R.J.
; SOARES, CARLOS R.J.

.
Molecular cloning and AlphaFold modeling of thyrotropin (ag-TSH) from the Amazonian fish Pirarucu (Arapaima gigas).
Bioinformatics and Biology Insights,
v. 17,
p. 1-13,
2023.
DOI:
10.1177/11779322231154148
Abstract:
Arapaima gigas, known as Pirarucu in Brazil, is one of the largest freshwater fish in the world. Some individuals could reach 3 m in length and weight up to 200 kg. Due to extinction risks and its economic value, the species has been a focus for preservation and reproduction studies. Thyrotropin (TSH) is a glycoprotein hormone formed by 2 subunits α and β whose main activity is related to the synthesis of thyroid hormones (THs)—T3 and T4. In this work, we present a combination of bioinformatics tools to identify Arapaima gigas βTSH (ag-βTSH), modeling its molecular structure and express the recombinant heterodimer form in mammalian cells. Using the combination of computational biology, based on genome-related information, in silico molecular cloning and modeling led to confirm results of the ag-βTSH sequence by reverse transcriptase-polymerase chain reaction (RT-PCR) and transient expression in human embryonic kidney (HEK293F) cells. Molecular cloning of ag-βTSH retrieved 146 amino acids with a signal peptide of 21 amino acid residues and 6 disulfide bonds. The sequence has a similarity to 39 fish species, ranging between 43.1% and 81.6%, whose domains are extremely conserved, such as cystine knot motif and N-glycosylation site. The Arapaima gigas thyrotropin (ag-TSH) model, solved by AlphaFold, was used in molecular dynamics simulations with Scleropages formosus receptor, providing similar values of free energy ΔGbind and ΔGPMF in comparison with Homo sapiens model. The recombinant expression in HEK293F cells reached a yield of 25 mg/L, characterized via chromatographic and physical-chemical techniques. This work shows that other Arapaima gigas proteins could be studied in a similar way, using the combination of these techniques, recovering more information from its genome and improving the reproduction and preservation of this prehistoric fish.
Palavras-Chave:
fishes;
thyroid hormones;
tsh;
molecules;
cloning;
toxicity
FREIRE, RENAN P.; HERNANDEZ-GONZALEZ, JORGE E.; LIMA, ELIANA R.; SUZUKI, MIRIAM F.; OLIVEIRA, JOAO E. de; TORATI, LUCAS S.; BARTOLINI, PAOLO; SOARES, CARLOS R.J.
Molecular cloning and AlphaFold modeling of thyrotropin (ag-TSH) from the Amazonian fish Pirarucu (Arapaima gigas).
Bioinformatics and Biology Insights,
v. 17,
p. 1-13,
2023.
DOI:
10.1177/11779322231154148.
Disponível em: http://repositorio.ipen.br/handle/123456789/33976. Acesso em: $DATA.
Esta referência é gerada automaticamente de acordo com as normas do estilo IPEN/SP (ABNT NBR 6023) e recomenda-se uma verificação final e ajustes caso necessário.
Como referenciar este item
-
LIMA, ELIANA R.
 ; CECCHI, CLAUDIA R.
; CECCHI, CLAUDIA R.
 ; HIGUTI, ELIZA
; HIGUTI, ELIZA
 ; JESUS, GUSTAVO P.P. de
; JESUS, GUSTAVO P.P. de
 ; GOMES, ALISSANDRA M.
; GOMES, ALISSANDRA M.
 ; ZACARIAS, ENIO A.
; ZACARIAS, ENIO A.
 ; BARTOLINI, PAOLO
; BARTOLINI, PAOLO
 ; PERONI, CIBELE N.
; PERONI, CIBELE N.

.
Optimization of mouse growth hormone plasmid DNA electrotransfer into tibialis cranialis muscle of "little" mice.
Molecules,
v. 25,
n. 21,
p. 1-9,
2020.
DOI:
10.3390/molecules25215034
Abstract:
Previous non-viral gene therapy was directed towards two animal models of dwarfism: Immunodeficient (lit/scid) and immunocompetent (lit/lit) dwarf mice. The former, based on hGH DNA administration into muscle, performed better, while the latter, a homologous model based on mGH DNA, was less efficient, though recommended as useful for pre-clinical assays. We have now improved the growth parameters aiming at a complete recovery of the lit/lit phenotype. Electrotransfer was based on three pulses of 375 V/cm of 25 ms each, after mGH-DNA administration into two sites of each non-exposed tibialis cranialis muscle. A 36-day bioassay, performed using 60-day old lit/lit mice, provided the highest GH circulatory levels we have ever obtained for GH non-viral gene therapy: 14.7 ± 3.7 ng mGH/mL. These levels, at the end of the experiment, were 8.5 ± 2.3 ng/mL, i.e., significantly higher than those of the positive control (4.5 ± 1.5 ng/mL). The catch-up growth reached 40.9% for body weight, 38.2% for body length and 82.6%–76.9% for femur length. The catch-up in terms of the mIGF-1 levels remained low, increasing from the previous value of 5.9% to the actual 8.5%. Although a complete phenotypic recovery was not obtained, it should be possible starting with much younger animals and/or increasing the number of injection sites.
Palavras-Chave:
gene therapy;
hormones;
dna;
muscles;
mice;
plasmids;
bioassay
LIMA, ELIANA R.; CECCHI, CLAUDIA R.; HIGUTI, ELIZA; JESUS, GUSTAVO P.P. de; GOMES, ALISSANDRA M.; ZACARIAS, ENIO A.; BARTOLINI, PAOLO; PERONI, CIBELE N.
Optimization of mouse growth hormone plasmid DNA electrotransfer into tibialis cranialis muscle of "little" mice.
Molecules,
v. 25,
n. 21,
p. 1-9,
2020.
DOI:
10.3390/molecules25215034.
Disponível em: http://200.136.52.105/handle/123456789/31768. Acesso em: $DATA.
Esta referência é gerada automaticamente de acordo com as normas do estilo IPEN/SP (ABNT NBR 6023) e recomenda-se uma verificação final e ajustes caso necessário.
Como referenciar este item
Buscar no repositório
Navegar
Minha conta
Visualizar
A pesquisa no RD utiliza os recursos de busca da maioria das bases de dados. No entanto algumas dicas podem auxiliar para obter um resultado mais pertinente.
✔ É possível efetuar a busca de um autor ou um termo em todo o RD, por meio do
Buscar no Repositório
, isto é, o termo solicitado será localizado em qualquer campo do RD. No entanto esse tipo de pesquisa não é recomendada a não ser que se deseje um resultado amplo e generalizado.
✔ A pesquisa apresentará melhor resultado selecionando um dos filtros disponíveis em
Navegar
✔ Os filtros disponíveis em
Navegar
tais como: Coleções, Ano de publicação, Títulos, Assuntos, Autores, Revista, Tipo de publicação são autoexplicativos. O filtro,
Autores IPEN
apresenta uma relação com os autores vinculados ao IPEN; o
ID Autor IPEN
diz respeito ao número único de identificação de cada autor constante no RD e sob o qual estão agrupados todos os seus trabalhos independente das variáveis do seu nome;
Tipo de acesso
diz respeito à acessibilidade do documento, isto é , sujeito as leis de direitos autorais, ID RT apresenta a relação dos relatórios técnicos, restritos para consulta das comunidades indicadas.

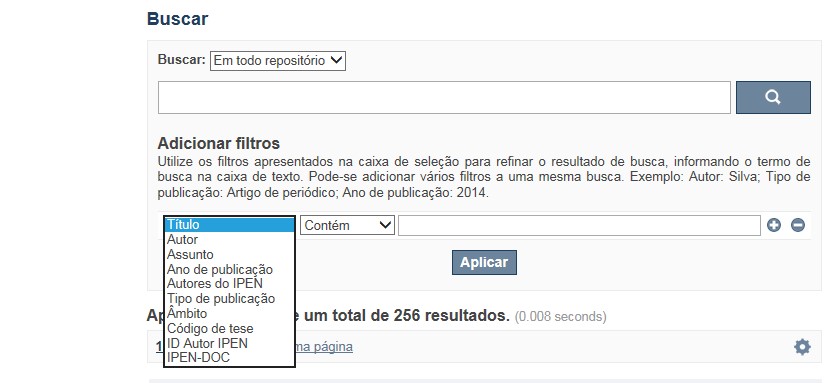
A opção
Busca avançada
utiliza os conectores da lógica boleana, é o melhor recurso para combinar chaves de busca e obter documentos relevantes à sua pesquisa, utilize os filtros apresentados na caixa de seleção para refinar o resultado de busca. Pode-se adicionar vários filtros a uma mesma busca.
Exemplo:
Buscar os artigos apresentados em um evento internacional de 2015, sobre loss of coolant, do autor Maprelian.
Autor: Maprelian
Título: loss of coolant
Tipo de publicação: Texto completo de evento
Ano de publicação: 2015
✔ Para indexação dos documentos é utilizado o Thesaurus do INIS, especializado na área nuclear e utilizado em todos os países membros da
International Atomic Energy Agency – IAEA
, por esse motivo, utilize os termos de busca de assunto em inglês; isto não exclui a busca livre por palavras, apenas o resultado pode não ser tão relevante ou pertinente.
✔ 95% do RD apresenta o texto completo do documento com livre acesso, para aqueles que apresentam o  significa que e o documento está sujeito as leis de direitos autorais, solicita-se nesses casos contatar a Biblioteca do IPEN,
bibl@ipen.br
.
significa que e o documento está sujeito as leis de direitos autorais, solicita-se nesses casos contatar a Biblioteca do IPEN,
bibl@ipen.br
.
✔ Ao efetuar a busca por um autor o RD apresentará uma relação de todos os trabalhos depositados no RD. No lado direito da tela são apresentados os coautores com o número de trabalhos produzidos em conjunto bem como os assuntos abordados e os respectivos anos de publicação agrupados.
✔ O RD disponibiliza um quadro estatístico de produtividade, onde é possível visualizar o número dos trabalhos agrupados por tipo de coleção, a medida que estão sendo depositados no RD.
✔ Na página inicial nas referências são sinalizados todos os autores IPEN, ao clicar nesse símbolo  será aberta uma nova página correspondente à aquele autor – trata-se da página do pesquisador.
será aberta uma nova página correspondente à aquele autor – trata-se da página do pesquisador.
✔ Na página do pesquisador, é possível verificar, as variações do nome, a relação de todos os trabalhos com texto completo bem como um quadro resumo numérico; há links para o Currículo Lattes e o Google Acadêmico ( quando esse for informado).
ATENÇÃO!
ESTE TEXTO "AJUDA" ESTÁ SUJEITO A ATUALIZAÇÕES CONSTANTES, A MEDIDA QUE NOVAS FUNCIONALIDADES E RECURSOS DE BUSCA FOREM SENDO DESENVOLVIDOS PELAS EQUIPES DA BIBLIOTECA E DA INFORMÁTICA.
O gerenciamento do Repositório está a cargo da Biblioteca do IPEN. Constam neste RI, até o presente momento 20.950 itens que tanto podem ser artigos de periódicos ou de eventos nacionais e internacionais, dissertações e teses, livros, capítulo de livros e relatórios técnicos. Para participar do RI-IPEN é necessário que pelo menos um dos autores tenha vínculo acadêmico ou funcional com o Instituto. Nesta primeira etapa de funcionamento do RI, a coleta das publicações é realizada periodicamente pela equipe da Biblioteca do IPEN, extraindo os dados das bases internacionais tais como a Web of Science, Scopus, INIS, SciElo além de verificar o Currículo Lattes. O RI-IPEN apresenta também um aspecto inovador no seu funcionamento. Por meio de metadados específicos ele está vinculado ao sistema de gerenciamento das atividades do Plano Diretor anual do IPEN (SIGEPI). Com o objetivo de fornecer dados numéricos para a elaboração dos indicadores da Produção Cientifica Institucional, disponibiliza uma tabela estatística registrando em tempo real a inserção de novos itens. Foi criado um metadado que contém um número único para cada integrante da comunidade científica do IPEN. Esse metadado se transformou em um filtro que ao ser acionado apresenta todos os trabalhos de um determinado autor independente das variáveis na forma de citação do seu nome.
A elaboração do projeto do RI do IPEN foi iniciado em novembro de 2013, colocado em operação interna em julho de 2014 e disponibilizado na Internet em junho de 2015. Utiliza o software livre Dspace, desenvolvido pelo Massachusetts Institute of Technology (MIT). Para descrição dos metadados adota o padrão Dublin Core. É compatível com o Protocolo de Arquivos Abertos (OAI) permitindo interoperabilidade com repositórios de âmbito nacional e internacional.
1. Portaria IPEN-CNEN/SP nº 387, que estabeleceu os princípios que nortearam a criação do RDI,
clique aqui.
2. A experiência do Instituto de Pesquisas Energéticas e Nucleares (IPEN-CNEN/SP) na criação de um Repositório Digital Institucional – RDI,
clique aqui.
O Repositório Digital do IPEN é um equipamento institucional de acesso aberto, criado com o objetivo de reunir, preservar, disponibilizar e conferir maior visibilidade à Produção Científica publicada pelo Instituto, desde sua criação em 1956.
Operando, inicialmente como uma base de dados referencial o Repositório foi disponibilizado na atual plataforma, em junho de 2015. No Repositório está disponível o acesso ao conteúdo digital de artigos de periódicos, eventos, nacionais e internacionais, livros, capítulos, dissertações, teses e relatórios técnicos.
A elaboração do projeto do RI do IPEN foi iniciado em novembro de 2013, colocado em operação interna em julho de 2014 e disponibilizado na Internet em junho de 2015. Utiliza o software livre Dspace, desenvolvido pelo Massachusetts Institute of Technology (MIT). Para descrição dos metadados adota o padrão Dublin Core. É compatível com o Protocolo de Arquivos Abertos (OAI) permitindo interoperabilidade com repositórios de âmbito nacional e internacional.
O gerenciamento do Repositório está a cargo da Biblioteca do IPEN. Constam neste RI, até o presente momento 20.950 itens que tanto podem ser artigos de periódicos ou de eventos nacionais e internacionais, dissertações e teses, livros, capítulo de livros e relatórios técnicos. Para participar do RI-IPEN é necessário que pelo menos um dos autores tenha vínculo acadêmico ou funcional com o Instituto. Nesta primeira etapa de funcionamento do RI, a coleta das publicações é realizada periodicamente pela equipe da Biblioteca do IPEN, extraindo os dados das bases internacionais tais como a Web of Science, Scopus, INIS, SciElo além de verificar o Currículo Lattes. O RI-IPEN apresenta também um aspecto inovador no seu funcionamento. Por meio de metadados específicos ele está vinculado ao sistema de gerenciamento das atividades do Plano Diretor anual do IPEN (SIGEPI). Com o objetivo de fornecer dados numéricos para a elaboração dos indicadores da Produção Cientifica Institucional, disponibiliza uma tabela estatística registrando em tempo real a inserção de novos itens. Foi criado um metadado que contém um número único para cada integrante da comunidade científica do IPEN. Esse metadado se transformou em um filtro que ao ser acionado apresenta todos os trabalhos de um determinado autor independente das variáveis na forma de citação do seu nome.


![]() significa que e o documento está sujeito as leis de direitos autorais, solicita-se nesses casos contatar a Biblioteca do IPEN,
bibl@ipen.br
.
significa que e o documento está sujeito as leis de direitos autorais, solicita-se nesses casos contatar a Biblioteca do IPEN,
bibl@ipen.br
.![]() será aberta uma nova página correspondente à aquele autor – trata-se da página do pesquisador.
será aberta uma nova página correspondente à aquele autor – trata-se da página do pesquisador.